來回顧一下短短的綜合分析篇學了什麼吧~
RNA-Sick@Day16 > 基因代號進得去,生物意義出得來,GO 發大財|基因本體論富集分析 feat. Gene Ontology (上)
RNA-Sick@Day17 > 基因代號進得去,生物意義出得來,GO 發大財|基因本體論富集分析 feat. Panther classification system (中)
RNA-Sick@Day18 > 基因代號進得去,生物意義出得來,GO 發大財|基因本體論富集分析 feat. REViGO (下)
RNA-Sick@Day19 > 快用你那無敵的非監督機器學習想想辦法吧|依據表現量特徵將基因分群 feat. MAPMAN (上)
RNA-Sick@Day20 > 快用你那無敵的非監督機器學習想想辦法吧|依據表現量特徵將基因分群 feat. K-means clustering (下)
RNA-Sick@Day21 > 是擅長畫 Pathway 的朋友啊|用程式來查京都基因與基因體百科全書 feat. KEGG REST API
RNA-Sick@Day22 > 誰能阻止少年專題生呢?他們聽不到|用程式上 NCBI 資料庫 feat. NCBI Entrez
RNA-Sick@Day23 > 基因調控什麼的最喜歡了|啟動子區段序列之順式作用元件分析 feat. PLACE database
RNA-Sick@Day24 > 關於資料視覺化,我想說的是|用 python 繪製充滿特色的圖表 feat. seaborn
RNA-Sick@Day25 > 我念 HMMER,但是我朋友都念 HMMER,聽說正解是HMMER|基於隱藏式馬可夫鍊的超強序列比對軟體 feat. HMMER3
HMMER 通常是在 genome-wide 的轉錄因子基因家族搜尋中使用,找出所有的轉錄音子基因家族成員之後,通常會在用 motif 軟體偵測一遍這些序列上還有沒有其他我們沒有檢查到的功能區段,這時候就會使用到 MEME 這軟體。
The MEME Suite 也是有自己的 MEME 宇宙的那種套裝軟體,但是我們通常只會用到 Motif Discovery 的功能。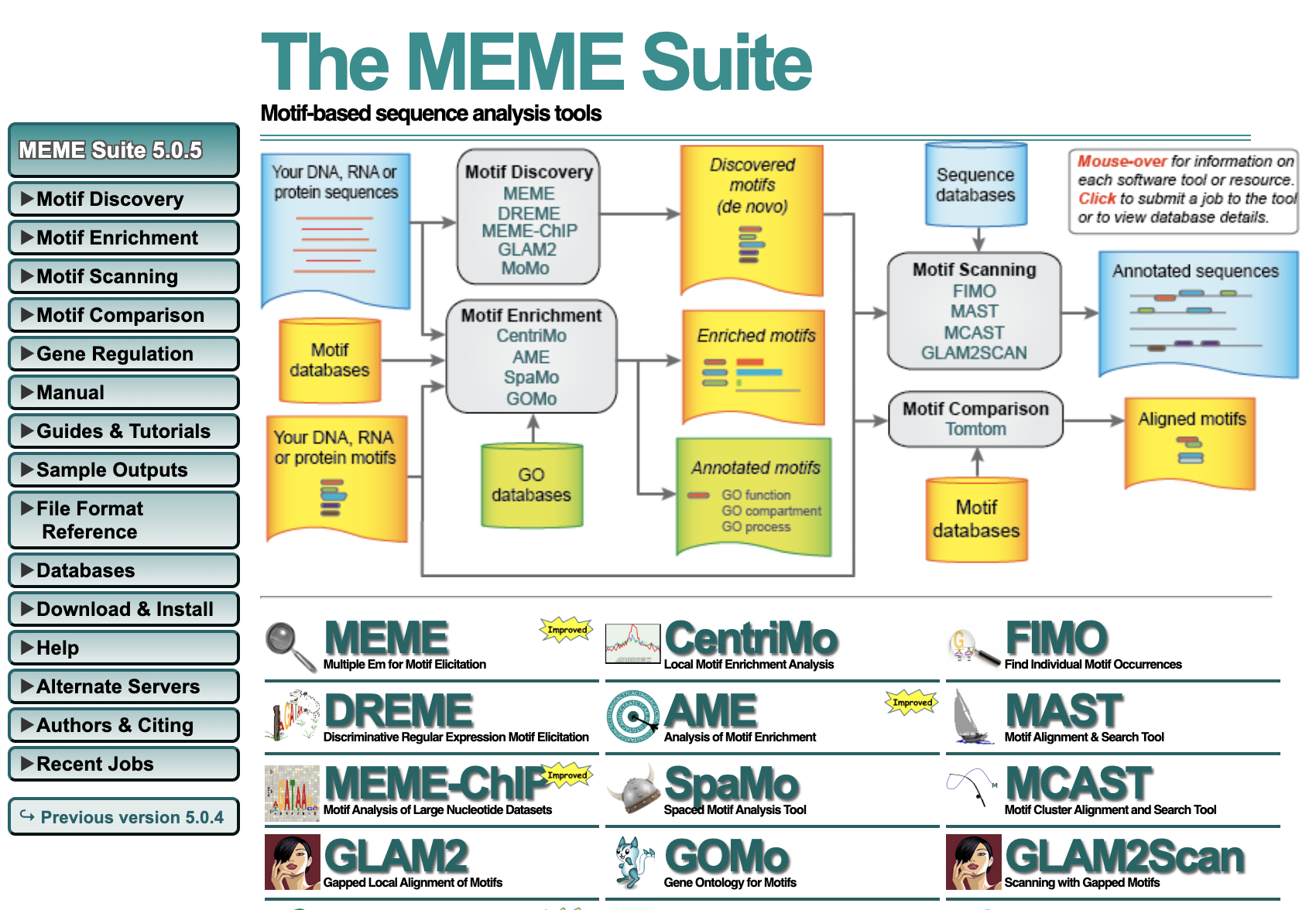
MEME 的中心思想,就是不需要資料庫累積的背景知識,直接由輸入的每一條序列來看,其中是否有出現很眼熟的 pattern,達到一定門檻的話就會被認作是一個 motif 啦!上傳序列之後有一些基本參數要設定,其中 advanced 的參數建議也要設定一下,比較重要的大概就是一段序列中可不可以出現重複的 motif、最大的 motif 可以多長、總共要找到多少個 motif,這三個對結果的影響頗大,要畫出好的圖可能需要下一番功夫調教合理的參數,因此是可以從分析結果看出研究者是隨便丟序列隨便拿圖來講、還是真的有概念要找出生物意義~
輸出結果會自動被丟進 MEME Suite 中的 MAST,從 MAST 的結果最方便作為後續的研究繪圖調整~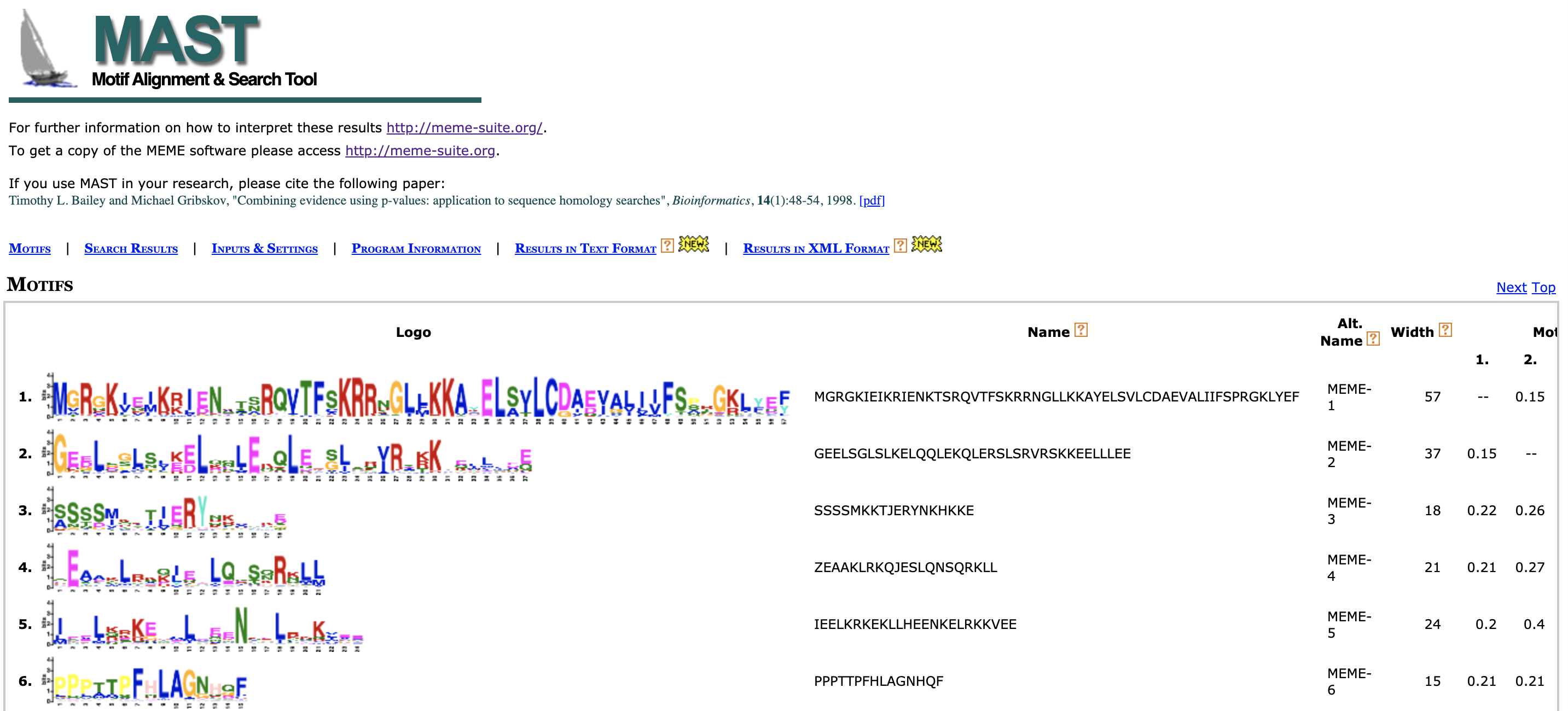
輸出的網頁介面可以進行互動式的序列檢視,十分方便,但是 MEME 存取我們上傳的分析有時間限制,建議盡快另存網頁下來,後續檢視與近一步分析比較方便!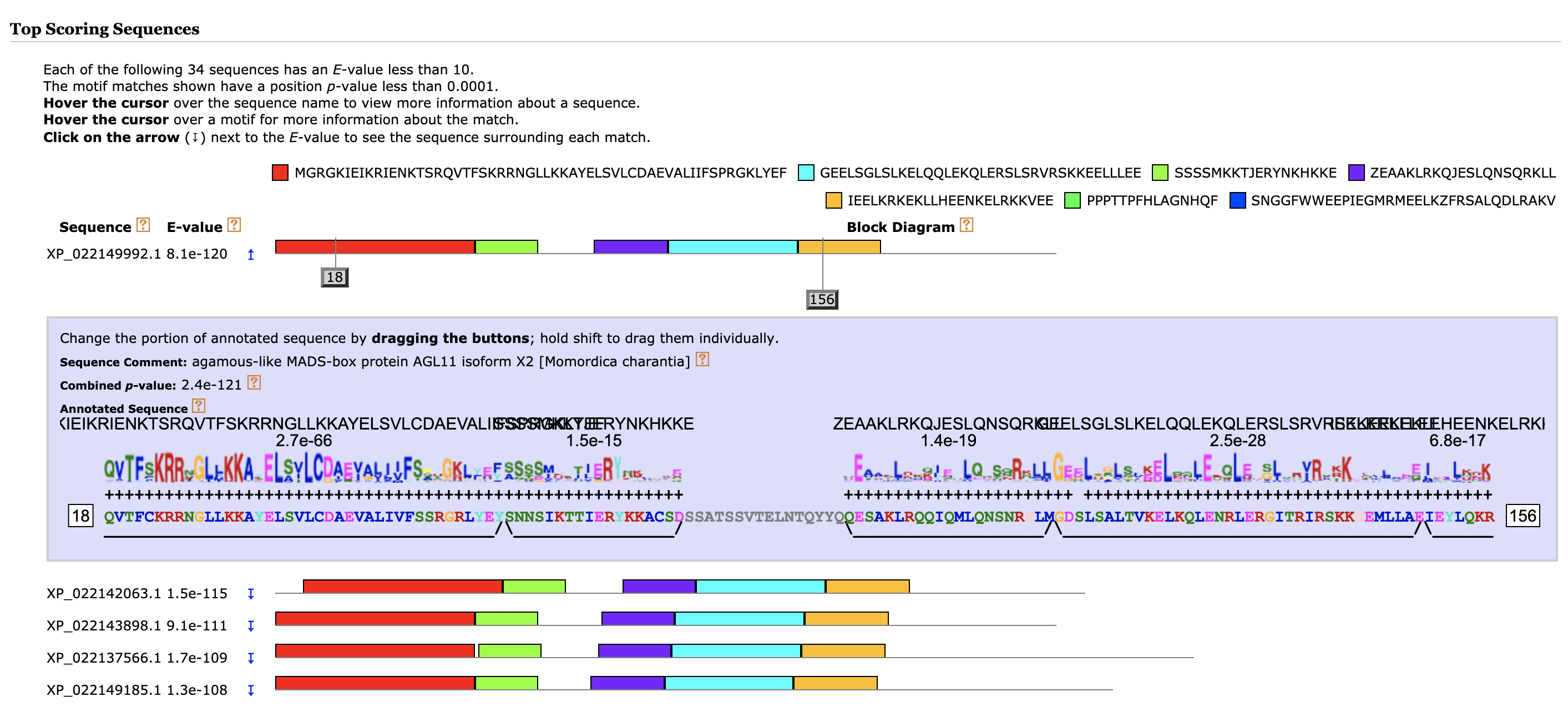
偵測完之後,每一段 motif 都只有一個代號,建議還是要透過序列比對來指定出究竟哪一段是已知的功能區段~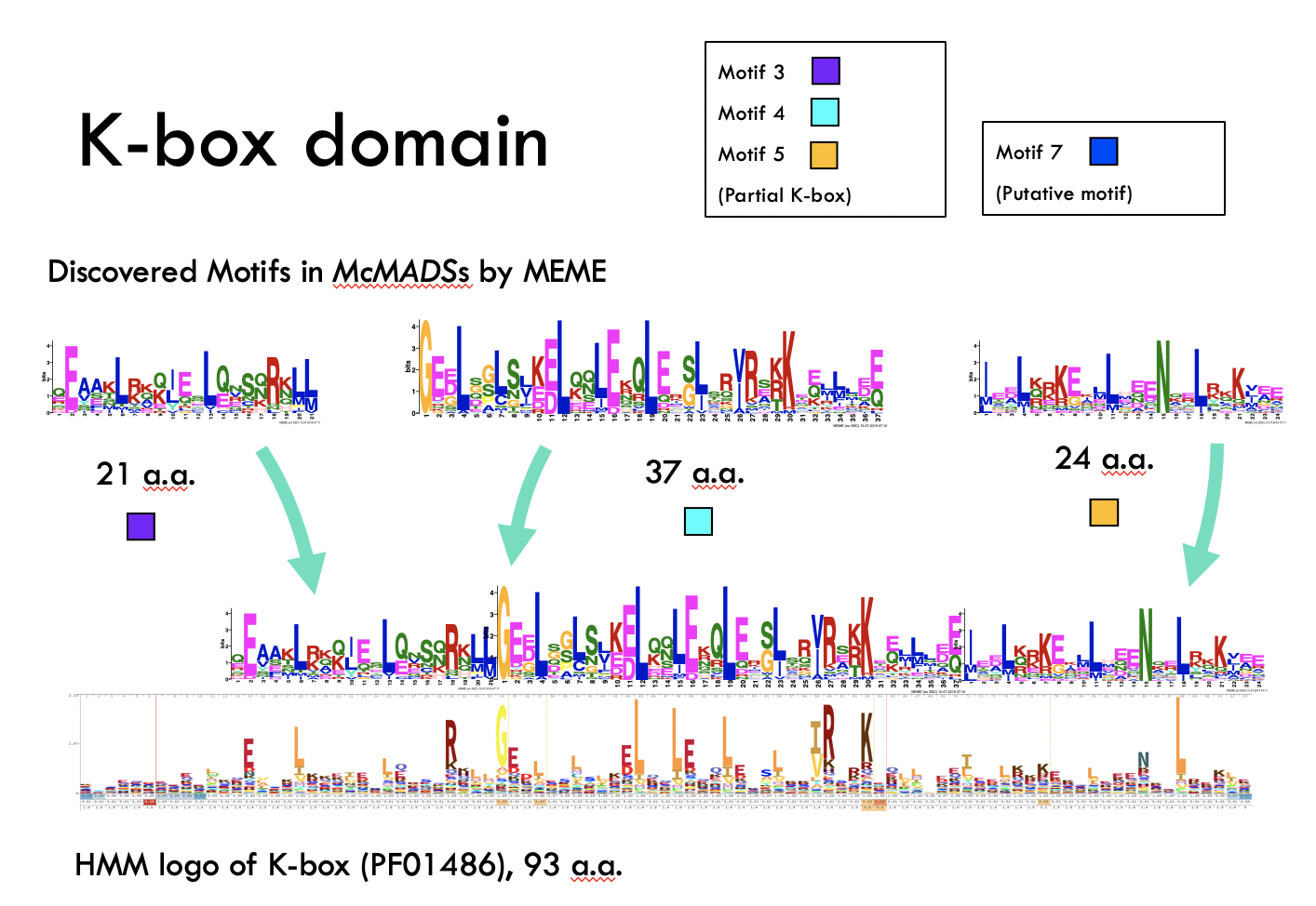
如果有什麼實際使用的問題,或是想要知道調教參數的密技,請留言告訴我~
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
